人類的DNA是由鹼基(ATCG)組成,人類DNA genome 大小3 x 10^9 bp (30億),DNA的資料量實在是很大,在過去要花很多錢才能夠分析,逐年發展,現在的科技已經可以用較為親民的價格分析所有的基因。
全基因定序(whole genome sequencing, WGS)和全外顯子定序(whole exome sequencing, WES)的差異在,會產生的蛋白的基因叫做外顯子也就是Protein-coding sequences,大概佔全部基因的1.5%,WES就只有分析會產生的蛋白的基因而已,價錢也較便宜,但WES已經是很強大的診斷工具,WGS or WES可以診斷出過去無法診斷的疾病,醫學的趨勢,進而達到精準診斷精準治療的需求。
人類DNA genome 大小3 x 10^9 bp (30億)
Protein-coding sequences 大約占整個基因體的1.5%
那之外的區域呢,有哪些種類?
那你該選哪種呢? 當然不用想更全面一定更好,所以WGS > WES?
當然沒有,$$$$ ,經費足夠一定選WGS,但$$當然永遠嫌不足,那如果你只要特定疾病,看特定基因,那就用Panels 就好了,因為可以針對你想要的區域特別去設計。
以下這張圖就能知道在各個DNA 測序的範圍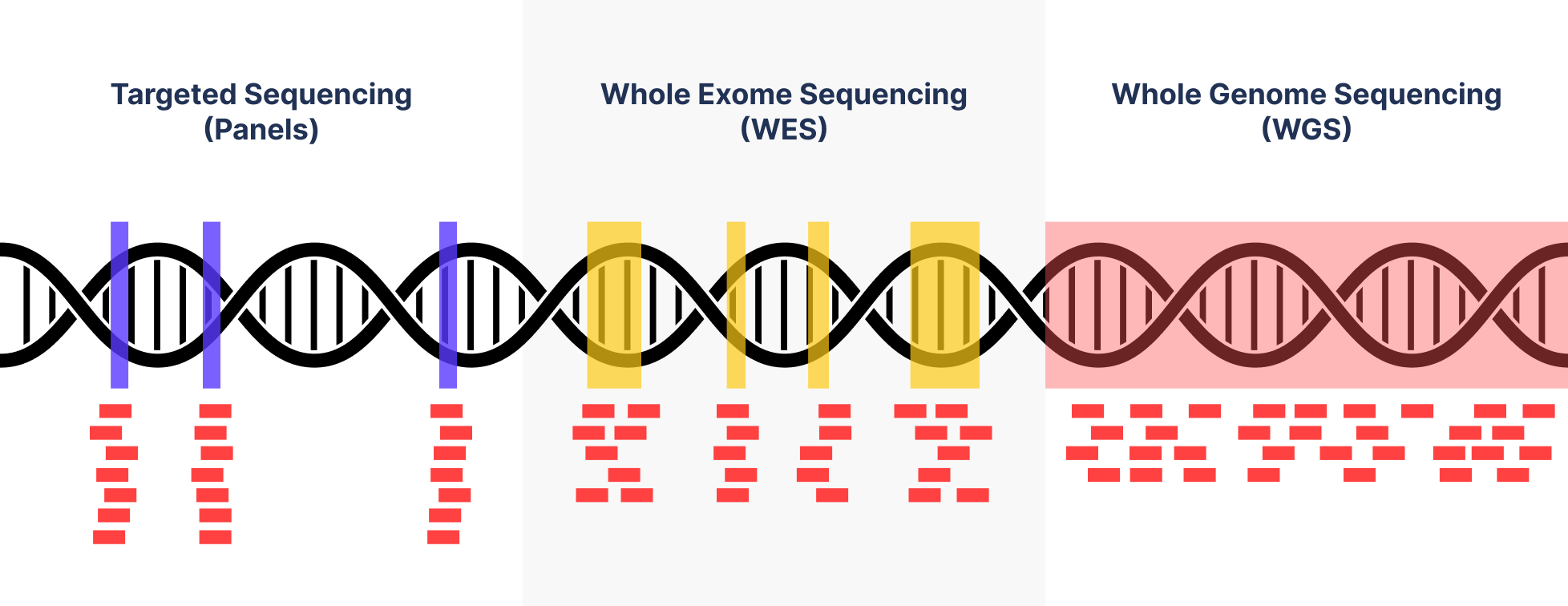
我們這次的系列會關注在NGS 次世代定序上面唷,所以接下來講這NGS 方面DNA測序的主要這三種差別
目前,NGS應用在DNA定序上的種類大致可以分為以下三類:
Whole Genome Sequencing (WGS):
中文:全基因組測序
描述:對整個人類基因組進行測序,但由於技術限制,某些區域難以測序,如高GC含量區域、大片段重複區域、染色體的中節和端粒。實際上,WGS覆蓋了基因組的大約95-98%區域。
Whole Exome Sequencing (WES):
中文:全外顯子測序
描述:僅對蛋白質編碼的區域(蛋白質編碼序列)進行測序,以降低成本和數據量。這些區域佔人類基因組的約1-2%,但包含大部分已知與疾病相關的變異。
Targeted Sequencing:
中文:目標定序
描述:針對特定區域或基因的測序,例如常見的癌症基因定序(癌症面板),主要用於研究與特定疾病或疾病類別相關的基因。這種方法可以根據研究興趣,有選擇地測序特定基因或區域,以節省成本和數據分析的時間。
總之,這三種DNA定序方法各有優勢,選擇取決於研究目的、預算和數據需求。
題外話:personal assembly
這是三代定序唷~~~
https://hkgp.org/tc/discover-genomics/genomics/%E5%85%A8%E5%9F%BA%E5%9B%A0%E7%B5%84%E6%B8%AC%E5%BA%8F/
WES WGS Panels: Which is the best NGS approach?
